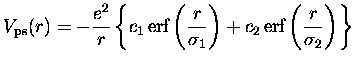
Dans cet annexe, nous allons donner les paramètres des pseudopotentiels locaux que nous avons utilisés : ceux-ci ont été choisis à la fois pour donner des résultats physiques sensés et pour être représentés de façon optimale sur la grille d'espace tridimensionnelle que nous avons utilisée pour représenter les variables spatiales.
Rappelons que dans le cas des agrégats de sodium nous utilisons des pseudopotentiels locaux, à symétrie sphérique, construits à partir de deux fonctions d'erreur :
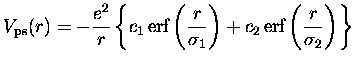
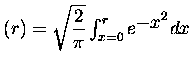 ) qui tend vers 1 pour
) qui tend vers 1 pour
La pseudodensité ionique
![]() est définie par
est définie par
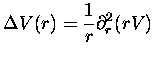
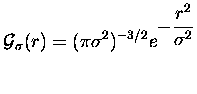
Pour caractériser ces pseudopotentiels, nous utilisons leur << force >>
![]() définie par
la mesure de la
différence du pseudopotentiel avec un potentiel coulombien :
définie par
la mesure de la
différence du pseudopotentiel avec un potentiel coulombien :
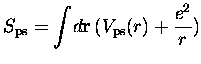
ce qui s'exprime en fonction de la pseudodensité par
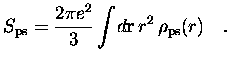
c'est à dire qu'il y a proportionnalité avec le rayon carré moyen de cette pseudodensité.
Pour la pseudodensité définie par E.2 cette relation donne
Le problème de la représentation sur réseau se pose alors.
L'expérience de travaux antérieurs de Reinhard et Suraud a montré que
des gaussiennes de largeur
![]() étaient représentées de façon optimale sur un réseau tridimensionnel à
maille la plus lâche possible à condition que l'espacement
étaient représentées de façon optimale sur un réseau tridimensionnel à
maille la plus lâche possible à condition que l'espacement ![]() des
points de ce réseau soit égal à la largeur à mi-hauteur
de
des
points de ce réseau soit égal à la largeur à mi-hauteur
de
![]() , soit
, soit
Il faut noter qu'il existe quatre coefficients pour nos pseudopotentiels
(deux largeurs et deux poids). Ils sont reliés par les
relations E.1, E.3, et E.4. Si on impose deux relations
de type E.4, comme nous l'avons fait pour imiter les
pseudopotentiels légèrement différents utilisés dans [38], tous les coefficients
sont définis, bien que le choix du pas de réseau soit assez arbitraire
(nous avons pris dans ce cas
![]() a
a![]() , ce qui était le
choix de [38], ce qui a fixé
, ce qui était le
choix de [38], ce qui a fixé
![]() , et pris
, et pris
![]() afin que la deuxième gaussienne soit représentée parfaitement sur le
réseau).
Dans un deuxième temps, vu que les résultats pour la position du pic
plasmon obtenus avec ces pseudopotentiels n'étaient pas satisfaisants,
nous avons pris un pas de réseau un peu plus serré et une force plus
grande pour les pseudopotentiels.
afin que la deuxième gaussienne soit représentée parfaitement sur le
réseau).
Dans un deuxième temps, vu que les résultats pour la position du pic
plasmon obtenus avec ces pseudopotentiels n'étaient pas satisfaisants,
nous avons pris un pas de réseau un peu plus serré et une force plus
grande pour les pseudopotentiels.
Les paramètres employés peuvent ainsi être résumés dans le tableau suivant :
| CAPS | plasmon mieux placé | ||
| 0,8 | 0,6366 | ||
| 0,96089 | 0,76472 | ||
| 1,92179 | 1,41421 | ||
| -0,90216 | -4,20716 | ||
| 1,90216 | 5,20716 | ||
|
|
38,907 | 49,97 |